La sustentabilidad urbana es, posiblemente, el problema ambiental más crítico que enfrenta la humanidad. Pero, ¿qué tan bien preparados están los planificadores urbanos para hacer frente a este problema, teniendo en cuenta que solo se basan en teorías e ideas exclusivas de las ciencias sociales en lugar de las ciencias biofísicas?
Tal y como ocurre en la naturaleza, las ciudades demandan recursos y productos como son los alimentos, agua y energía para poder vivir, ya obtenidos pasan por determinados procesos “fisiológicos” que producen sustancias y energía necesarias para mantener la vida, todo este concepto es el “Metabolismo Urbano”.
El Metabolismo Urbano ofrece un marco de trabajo holístico con el que puede analizar las entradas y salidas con respecto al medio biofísico que rodea a la ciudad, que demandan recursos como la naturaleza, sin embargo, en la naturaleza hay un metabolismo urbano circular, donde no se generan residuos, mientras que en las ciudades se agotan los recursos. Esto es porque las ciudades de hoy en día se basan en metabolismo lineales, extrayendo materias primas, fabricando productos para su consumo y desechándolos tras su uso, causando contaminación y la alta dependencia de recursos no renovables.
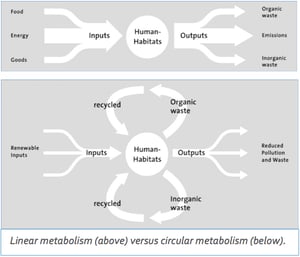
Con la aplicación adecuada de este concepto se pretende alcanzar un nuevo paradigma que transforme la gestion lineal e insostenible de los recursos en unos flujos que sean circulares. ¿Cómo alcanzar un metabolismo urbano circular?, básicamente consiste en gestionar los recursos a través de las ciudades, tomando en cuenta las salidas (residuos orgánicos e inorgánicos) como las entradas (reciclaje y recuperación) y que son capaces de volver al sistema productivo.
Un ejemplo es el tratamiento microbiológico en aguas residuales. Generalmente, el ciclo urbano del agua comienza con el agua ya potabilizada, que es transportada y entra de esta forma en los núcleos urbanos. Tras su utilización a lo largo de distintas actividades (por ejemplo, procesos industriales, uso doméstico, etc.) la calidad del agua disminuye de manera que se vierte como agua residual, abandonando así el sistema urbano. Por lo tanto debemos disponer de metodologías para conseguir agua de calidad, así como protocolos para valorar la efectividad de los tratamientos químios y biológicos empleados para su potabilización. (Ver estudio del tecnológico de monterrey)
Ahora bien, ¿cómo podemos asegurarnos de la calidad microbiológica del agua?, al momento de analizar cada microorganismos que pudiera estar presente en el agua no es muy práctico, es por esto que existen ciertos métodos generales que nos pueden señalar mediante la presencia de bacterias “indicadoras” de calidad sanitaria. Así podremos evaluar la calidad del agua, detectar la presencia de microorganismos en las aguas para usos especiales como en la industria farmacéutica, precisar la presencia de metabolitos microbianos, entre otros.
¿Qué procedimientos te recomiendo?, hoy te hablaré de dos métodos que te permitirán la identificación microbiana.
GENÓMICA
Para determinar la genómica de los microorganismos utilizamos la secuenciación, donde se determina el orden exacto de un genoma compuesto por pares de bases nucleicas, aquí es requerido una muestra de ÁCIDO NUCLEICO (ADN). Podemos caracterizar y monitorear comunidades microbianas, por alguno de los siguientes métodos:
- Secuenciar una región genómica específica (iSeq / MiSeq)
- Bacterias mediante Microbioma (región 16S)
- Hongos mediante Micobioma (región ITS)
- Secuenciar todo el genoma microbiano presente (NextSeq)
- Bacterias, hongos, virus, protozoarios, etc., (Shotgun sequencing)
PROTEÓMICA
Ahora bien para determinar proteínas ribosómicas el MALDI-TOF en Microbiología es el método adecuado para ti. El conjunto de los picos de masas constituye el espectro del microorganismo (huella peptídica) requiriendo una muestra de PROTEÍNA (aminoácidos).
La diferencia más grande entre ambas tecnologías es que MALDI-TOF es un método que requiere CULTIVO PREVIO (culture-dependent), por lo que se deben aislar los microorganismos, antes que la extracción de proteínas. En NGS, los métodos de 16S y shotgun son totalmente libres de cultivo (culture-independent) por lo que se puede interrogar una muestra ambiental directamente sin aislamiento previo.
Existen reportes en los que ambos métodos se usan complementarios, pues MALDI-TOF depende de que exista una base de datos de referencia para comparar la huella proteica del microorganismo, mientras que en NGS se puede ensamblar un genoma totalmente nuevo (de novo) sin referencia alguna.
¿Quieres saber más de estas dos técnicas y como aplicarlas en tu proyecto?
Si deseas saber más sobre el tema, comenta queremos saber de ti, para mayor contenido científico suscribete a nuestro blog y síguenos en nuestras redes sociales. Porque sabemos que tú puedes cambiar al mundo en Analitek…
Tu éxito es nuestra pasión











Comment