El SARS-CoV-2, el virus responsable de la pandemia de Covid-19, es un nuevo patógeno identificado por primera vez en China a fines de diciembre de 2019. Desde que se publicó el primer genoma del SARS-CoV-2 el 24 de enero, los investigadores han estado recopilando información rápidamente. sobre el virus para ayudar a informar las iniciativas de salud pública.
A fines de abril, se habían cargado más de 12,000 secuencias del genoma del SARS-CoV-2 en GISAID, una base de datos de acceso abierto que rastrea la evolución viral y la propagación en todo el mundo. Este número está muy por detrás de los más de tres millones de casos de infección detectados por las pruebas de reacción en cadena de la polimerasa (PCR), lo que destaca la dificultad para contener la propagación del virus y realizar un seguimiento de su secuencia genética a lo largo del tiempo.
Parte del problema es que el SARS-CoV-2 causa un amplio espectro de síntomas clínicos, desde muy leves hasta críticos. Para que sean eficaces, las medidas de control y prevención de infecciones deben tener en cuenta todos los tipos de transmisión, incluso por parte de personas asintomáticas o presintomáticas. De manera similar, otras infecciones podrían afectar el curso de Covid-19; el conocimiento de estos también podría ayudar a identificar a los pacientes que tienen un mayor riesgo de enfermedad grave.
Ahora bien, las pruebas de PCR actuales solo determinan la presencia del virus. “No dice nada sobre la secuencia genética del virus, la presencia de coinfección ni ninguna información sobre la respuesta inmunitaria del paciente”, Gary Schroth, distinguido científico y vicepresidente de Illumina nos menciona; “Un sistema de secuenciación de próxima generación (NGS) que respalde la PCR es realmente importante para garantizar que la prueba siga funcionando bien y así obtener esta valiosa información”.
Para ayudar a los investigadores a trabajar hacia estos objetivos, Illumina ha ideado dos métodos para secuenciar el SARS-CoV-2 a partir de muestras clínicas: un flujo de trabajo basado en la metagenómica shotgun y otro en el enriquecimiento del target.
Shotgun metagenomics workflow
La metagenómica shotgun es un método NGS de alto rendimiento que identifica y genotipa todas las comunidades microbianas en una muestra sin conocimiento previo de qué microbios están presentes. El examen completo de todos los organismos en una muestra es crucial si se van a identificar nuevos patógenos pandémicos, como el SARSCoV-2.
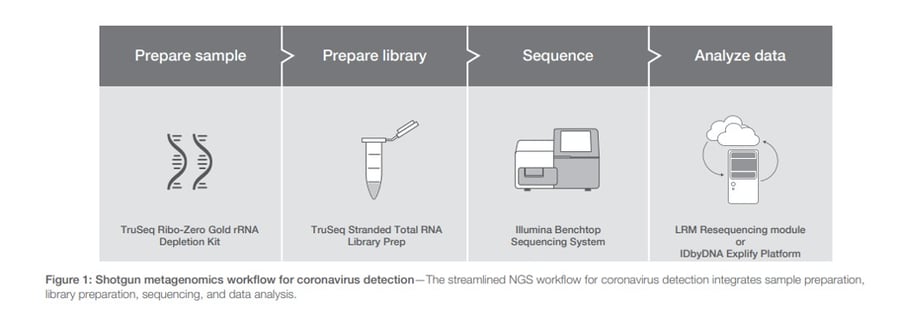
Comprehensive workflow for detecting coronavirus using Illumina benchtop systems Illumina
El flujo de trabajo de metagenómica shotgun de Illumina integra la preparación de muestras, la preparación de bibliotecas, la secuenciación y el análisis, ya sea utilizando datos locales o con herramientas basadas en la nube. Para este último, Illumina se ha asociado con la empresa de metagenómica IDbyDNA; los clientes pueden acceder a Explify Platform, una base de datos patentada de secuencias de referencia de ADN y ARN seleccionadas para identificar más de 35 000 virus y 13 000 bacterias.
La técnica de la escopeta puede determinar si los pacientes están coinfectados con otros virus, como el de la influenza o bacterias, lo que podría ser útil para informar futuras decisiones de tratamiento y predecir los resultados de los pacientes. El conocimiento de la composición genética del virus también permite a los investigadores comprender cómo está evolucionando el SARSCoV-2 para monitorear la transmisión y garantizar que las pruebas de diagnóstico y los tratamientos dirigidos a los productos del genoma viral sigan siendo efectivos. También proporciona información sobre la respuesta de los pacientes a la infección, esto podría ayudar a los investigadores a comprender cómo el SARSCoV-2 causa enfermedades y por qué afecta a algunas personas más que a otras.
Target enrichment workflow
El enriquecimiento de objetivos permite a los investigadores secuenciar de manera confiable genes específicos en una muestra, lo que reduce la cantidad de datos que requieren análisis.
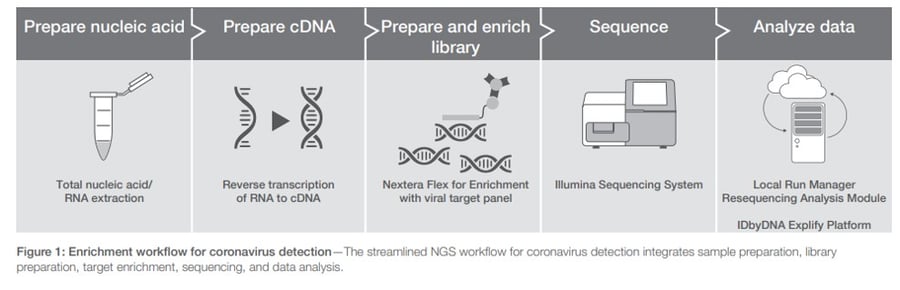 Enrichment workflow for detecting coronavirus using Illumina NGS systems Illumina.
Enrichment workflow for detecting coronavirus using Illumina NGS systems Illumina.
El flujo de trabajo de enriquecimiento de Illumina detecta cepas de coronavirus con una precisión equivalente a la secuenciación Shotgun. Este flujo de trabajo combina Nextera Flex for Enrichment de Illumina, que genera bibliotecas NGS específicas, y un panel de oligovirus respiratorios recientemente disponible, que captura y permite la resecuenciación de más de 40 patógenos.
El panel de enriquecimiento viral detecta mutaciones genómicas en diferentes muestras, lo que ayuda a definir la epidemiología de la transmisión. Los dos flujos de trabajo difieren en la profundidad de secuenciación (número de lecturas por muestra) requerida.
Estos dos flujos de trabajo complementarios destacan cómo se puede usar NGS para descubrir nuevos patógenos como el SARSCoV-2 y se puede usar para la vigilancia, determinando cómo está evolucionando el virus y las consecuencias de esos cambios. Los flujos de trabajo se pueden realizar junto con los métodos de prueba tradicionales y se pueden utilizar para optimizar las estrategias de control de infecciones. Es por esto que en Analitek life cuentas con nuestro apoyo para adentrarte a la secuenciación de próxima generación.
Si deseas conocer más de esta tecnología te invito a escribirnos al correo genomics@analitel.com y darte una asesoría gratuita.
Hoy más que siempre estamos contigo, en Analitek life…
Damos vida a la vida







Comment